聚类概念
- 属于无监督学习(无标签)
- 顾名思义,就是将相似的东西分到一组
- 难点在于如何评估聚类效果以及调整参数
基本概念
-
K代表最后需要得到的簇个数(比如二分类K=2)
-
一个簇的质心计算:采用均值,即每个特征向量取平均值
-
距离度量:经常使用欧几里得距离和余弦相似度(高维特征最好先执行一次标准化)
-
优化目标:实现$min\sum_{i=1}^{k}\sum_{x∈C_i}dist(C_i,x)^2$
优缺点
优点:
- 算法简单、快速
- 适合常规数据集(就是一坨一坨的那种,没有特定形状)
缺点:
- K值难以确定(一般是遍历K值取Inertia的拐点)
- 算法复杂度与样本呈线性关系(一次迭代)
- 很难发现任意形状的簇,比如二维特征的样本,一个圆环套一个球的形状KMeans就很难检测出来。

算法流程
- 首先随机选取K个样本作为K个簇的中心(在KMeans++中,使用最大距离进行初始化)
- KMeans++逐个选取K个簇中心,且离其它簇中心越远的样本点越有可能被选为下一个簇中心
- 将每个样本分配到K个簇中,具体选择哪个簇取决于该样本和K个簇的距离,选择距离最近的簇(分配样本)
- 此时所有样本都分配到对应的簇,在每个簇中更新簇中心。新的簇中心采用简单的均值计算(更新簇中心)
- 回到第二步再次分配样本,直到达到迭代次数上限或损失指标小于某个阈值(损失指标有inertia/轮廓系数)
KMeans代码实现
import numpy as np
class KMeans:
def __init__(self,data,num_clusters):
self.data = data
self.num_clusters = num_clusters
def train(self,max_iterations):
# 1.choose k centers
centroids = KMeans.centroids_init(self.data,self.num_clusters)
# 2.begin train
num_examples = self.data.shape[0]
closest_centroids_ids = np.empty((num_examples,1))
for _ in range(max_iterations):
# 3.get closest center id
closest_centroids_ids = KMeans.centroids_find_closest(self.data,centroids)
# 4.update k centers
centroids = KMeans.centroids_compute(self.data,closest_centroids_ids,self.num_clusters)
return centroids,closest_centroids_ids
@staticmethod
def centroids_init(self,data,num_clusters):
num_examples = data.shape[0]
random_ids = np.random.permutation(num_examples)
centroids = data[random_ids[:num_clusters],:]
return centroids
@staticmethod
def centroids_find_closest(self,data,entroids):
num_examples = data.shape[0]
num_centroids = centroids.shape[0]
closest_centroids_ids = np.zeros((num_examlpes,1))
for example_index in range(num_examples):
distance = np.zeros(num_centroids,1)
for centroid_index in range(num_centroids):
distance_diff = data[example_index,:] - centroids[centroid_index,:]
distance[centroid_index] = np.sum(distance_diff**2)
closest_centroids_ids[example_index] = np.argmin(distance)
return closest_centroids_ids
@staticmethod
def centroids_compute(self.data,closest_centroids_ids,self.num_clusters):
num_features = data.shape[1]
centroids = np.zeros((num_clusters,num_features))
for centroid_id in range(num_clusters):
closest_ids = closest_centroids_ids == centroid_id
centroids[centroid_ids] = np.mean(data[closest_ids.flatten(),:],axis=0)
return centroidssklearn
接下来介绍sklearn库中的KMeans实现.
文档见sklearn.cluster.KMeans — scikit-learn 1.2.2 documentation
Attributes

__init__
n_clusters: 簇个数Krandom_state: 随机数种子init: K个簇中心的初始化方式n_init: 算法执行次数,取结果最好的一次max_iter: 单次算法迭代次数(分配簇/更新簇)
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=2,random_state=0,init='k-means++',n_init=10,max_iter=300)fit
kmeans.fit(X) # input X
kmeans.cluster_centers_ # center coordinate
kmeans.label_ # every sample's label
kmeans.inertia # losspred
kmeans.predict(New_X)
kmeans.label_tranform
Transform(): Method using these calculated parameters apply the transformation to a particular dataset.
返回值shape : (num_examples,num_clusters) 即每个样本和每个类的距离 通过这个方法也可以得到predict结果(argmin)
from sklearn.preprocessing import StandardScaler
sc = StandardScaler()
sc.fit_tranform(X_train)
sc.tranform(X_test) # 一定是tranform 保证train和test使用同一个转化标准可视化
# draw cluster points
def plot_data(feature,snr,label):
df = pd.DataFrame(dict(x=snr, y=feature, color=label))
fig, ax = plt.subplots()
colors = {0:'red', 1:'blue'}
ax.scatter(df['x'], df['y'], c=df['color'].apply(lambda x: colors[x]))
plt.show()
# draw cluster centers
def plot_centriods(snr,centroids,weights=None,circle_color='g',cross_color='k'):
if weights is not None:
centroids = centroids[weights>weights.max()/10]
plt.scatter(snr,centroids[:],
marker='o',s=30,linewidths=8,
color=circle_color,zorder=10,alpha=0.9)
plt.scatter(snr,centroids[:],
marker='x',s=50,linewidths=50,
color=circle_color,zorder=11,alpha=1)评估指标
inertia指标
inertia : 每个样本距离质心的距离
score方法的结果是inertia的负值(绝对值越大分数越小)
kmeans.inertia_
X_dist = kmeans.transform(X)
np.sum(X_dist[np.arange(len(X_dist)),kmeans.labels_]**2) # inertia计算
kmeans.score(X) == -1*kmeans.inertia_轮廓系数
某个样本的轮廓系数定义为:
$$
s = \frac {disMean_{out} - disMean_{in}}{max(disMean_{out}-disMean_{in})}
$$
其中$disMean_{in}$为该点与本类其他点的平均距离,$disMean_{out}$为该点与非本类点的平均距离。该值取值范围为[−1,1], 越接近1则说明分类越优秀。在sklearn中函数 silhouette_score()计算所有点的平均轮廓系数,而silhouette_samples()返回每个点的轮廓系数。
K值选取(拐点法)
kmeans_per_k = [KMeans(n_clusters=k).fit(X) for k in range(1,10)]
inertias = [model.inertia_ for model in kmeans_per_k]
# draw fig
plt.figure(figsize=(8,4))
plt.plot(range(1,10),inertias,'bo-')
plt.show()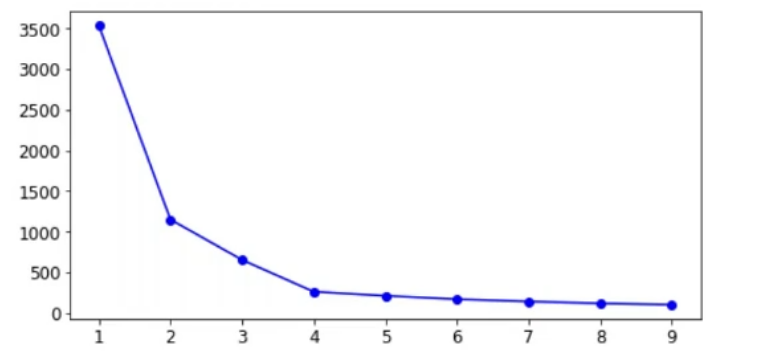
半监督学习
KMeans也可以用于半监督学习。
比如在一个少样本训练场景下,相比于随机选取50个训练样本打标加入训练这种方式,可以先使用聚类挑出50个簇中心,每个簇选取一个样本作为典型样本;用这些样本打标训练,最后测试出来的acc会更高。除此之外,还可以通过标签传播(同一个簇内的标签一致)的方式扩大训练集。

评论